KT는 유클라우드비즈 서비스에서 인간 유전체 분석을 위한 플랫폼을 서비스하고 있다. KT의 유전체 분석 클라우드 서비스는 얼마전 도커 컨테이너 기반으로 개편됐다.
김진식 KT 미래사업개발단 차장은 29일 열린 ‘클라우드 프론티어2015’에서 클라우드 상에서 도커에 기반해 유전체 빅데이터를 분석한 사례를 공유했다.
KT의 게놈클라우드(GenomeCloud)는 유전체 분석에 특화된 대용량 정보 처리 서비스 플랫폼이다. 이중 개인 맞춤형 유전정보를 해석해 제공하는 바티컴(Vaticum)이란 연계 플랫폼이 있다. 질병 민감도, 암 발병확률 등을 알아낼 수 있다.
바티컴은 30억쌍의 DNA 서열정보를 분석하고, 4가지 이상의 툴로 10여단계를 거치는 복잡한 연산으로 서열 정보를 도출한다. 이때 참조하는 데이터베이스가 10여종 이상이고, 매핑하는 생물학 정보가 2억건 이상이다. 그렇게 유전체를 분석하는 소프트웨어 프로세스인 ‘파이프라인’이 상품별로 생성되고 업데이트 된다.
바티컴은 도커 컨테이너를 기반으로 구축됐다. 여러 파이프라인을 제각기 다른 환경에서 동시에 실행하고, 형상 관리도 편하기 때문에 활용하게 됐다는 설명이다.

김진식 차장은 “염기서열을 알아내는 과정에서 좀 더 들어가보면, 여러 툴이 다양한 옵션을 갖고 여러번의 단계를 거쳐 프로세싱하게 되는데 목적마다 조금씩 다르다”며 “어느 유전자가 어떤 변이를 일으킨다는 식의 다양한 연구성과가 DB화돼 참조하고, 매핑하는 작업이 이뤄져야 한다”
그는 “도커란, 리눅스 기반의 경리형 가상 독립환경을 제공하는 오픈 플랫폼으로, 애플리케이션 레벨의 가상화라고 볼 수 있다”며 “가상머신(VM)은 서버에 여러 대몬을 돌려서 복잡하지만, 컨테이너는 내부의 프로세서와 함께 실행됐다가 완료되면 종료된다”고 설명했다.
그는 이미지 버전 관리를 쉽게 할 수 있고, 사설 저장소를 만들 수 있다는 점을 도커의 또 다른 장점으로 꼽았다. 그중에서 이미지 버전관리를 가장 큰 장점으로 지목했다.
그는 “도커는 이미지 버전관리가 필요한 경우 유용한데, 버티컴에 VM을 쓰면 여러 파이프라인 유형을 분석별로 VM 스냅샷으로 만들고, 전부 다 직접 버전 관리해야 했을 것”이라며 “도커는 관리할 체계를 갖고 있어서 훨씬 간단하다”고 말했다.
버티컴은 파이프라인 전용 컨테이너 다수로 이뤄져 있다. 파이프라인 실행을 위한 잡이 있으면, 관련 컨테이너가 작동했다가 작업 종료 후 꺼진다. 유전체 분석의 경우 단일 데이터 규모가 큰 반면, 서버끼리 정보를 주고 받을 일이 없어 IO 부담이 없다.
관련기사
- "고효율 클라우드, 플래시스토리지가 이끈다"2015.10.29
- KT, 미국에도 클라우드 인프라 구축한다2015.10.29
- KT, IoT기기-앱 개발자에 손짓2015.10.29
- KT, 5G 연계한 클라우드 전략 확대한다2015.10.29
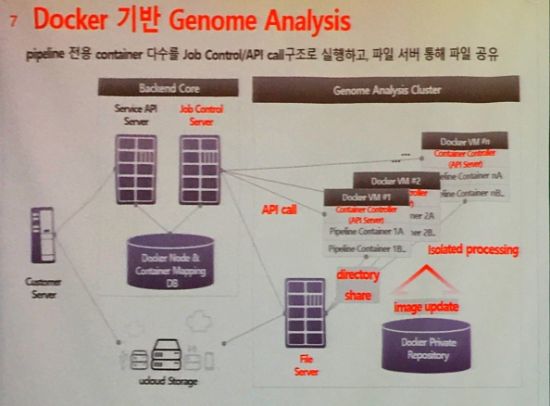
그는 “도커가 필요한 이미지를 찾아서 인풋을 가지고 실행해서 끝나면 돌려주는 형태”라며 “대규모의 잡을 동시수행하고, 유전자 데이터는 하나의 파일서버를 공유하며, 사설 저장소에 버전 이미지를 넣어 관리한다”고 밝혔다.
이어 “컨테이너 한대에서 하나의 파이프라인을 처리하는데, 한대에서 여러 파이프라인을 돌리는 식도 도입할 수 있을 거라 본다”며 “처음 도커를 우연히 접하고 실행한 뒤 너무 편해서, 바로 유전체 분석에 딱 어울리는 툴이라고 생각했다”고 말했다.