인간 유전체 정보를 모두 해석하는 '인간 지놈 프로젝트'는 1990년 시작되어 13년 만인 2003년 마무리되었다. 하지만 당시 기술의 한계로 다 해독하지 못 하고 남은 염기 서열들이 일부 있었다.
이후 세계의 과학자들이 지놈 정보를 지속적으로 업데이트했지만, 20년이 지나도록 전체 지놈의 8%는 미지의 영역으로 남아 있었다.
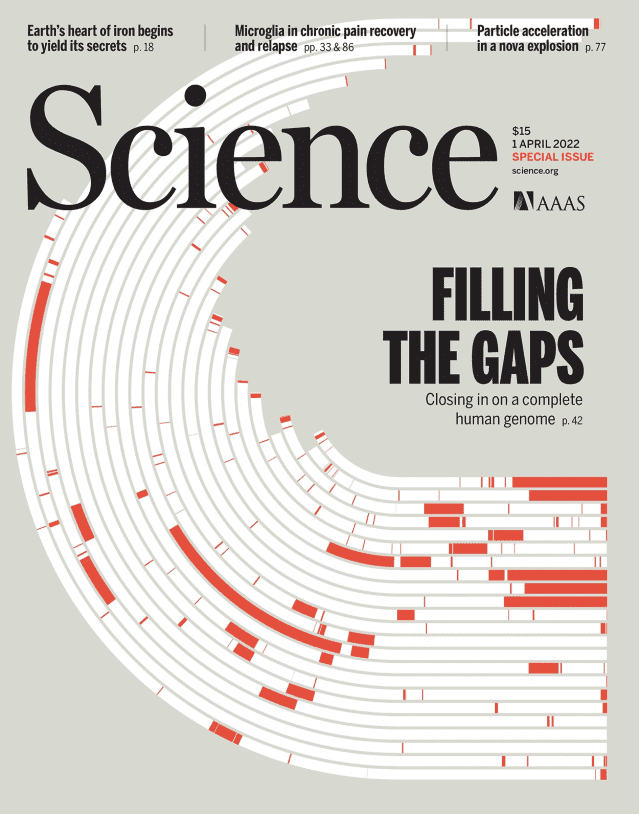
미국 인간지놈연구소(NHGRI)와 캘리포니아주립 산타크루즈대학 등 세계 과학자들로 구성된 '텔로미어-투-텔로미어(T2T) 컨소시움'은 지난 3년 간 이같은 정보의 빈틈을 메워 완전판 지놈 지도를 만드는 작업을 벌였다.
마침내 이 작업이 마무리됨에 따라 새로운 지놈 정보와 이를 통해 새롭게 발견한 사실들, 염기 서열 해독에 쓰인 신기술 등을 담은 여러 편의 논문이 31일(현지시간) 학술지 '사이언스'와 '네이처 메소즈(Nature Methods)'에 일제히 실렸다.
새로 발견된 유전체 정보를 바탕으로 질병 치료에 관련된 유전자를 찾아 치료법을 개발하는 과정이 빨라질 것으로 연구진은 기대했다.
■ 해독 어려웠던 '쓰레기 DNA' 영역 이해 도전
T2T 컨소시움은 염색체의 한쪽 끝 부분에 있는 텔로미어에서 다른 쪽 텔로미어까지의 염기 서열을 모두 밝히겠다는 의미로 지은 이름이다.
이들이 발표한 새 준거 유전체 'T2T-CHM13'은 30억 개 이상의 염기쌍과 2만 개 가까운 단백질 코딩 유전자로 구성돼 있다. 2003년 지놈 프로젝트의 결과물인 'GRCh 38'에 비해 2억 개의 염기쌍이 추가됐다. 또 연구진은 2천 개의 단백질 코딩 유전자를 새로 발견했으며, 이중 115개가 특성을 발현하는 유전자였다.
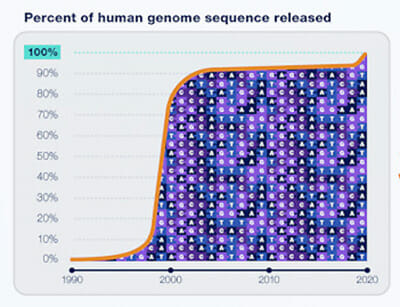
이번 연구 결과에는 그간 기술적 문제로 해독이 어려웠던 염기 서열들이 상당 부분을차지해 주목된다. 염색체 끝부분의 텔로미어나 세포 분할에 관여하는 센트로미어 등 염색체 주요 부분에는 같은 내용이 매우 길게 반복되는 염기서열이 있다. 이들은 단백질 코딩이 일어나지 않는 영역이라 생명 활동에 도움을 주지 않는 '쓰레기 DNA'로 간주되기도 했다.
하지만 최근 이들 영역이 세포 기능에 중요한 역할을 하며, 잘못될 경우 암의 원인이 될 수도 있다는 점 등이 드러나고 있다. 이런 가운데 극도로 긴 염기 서열을 읽어내는 '나노포어 시퀀싱' 등 새 기술을 활용해 이들 영역의 유전 정보를 밝힘에 따라, 향후 생물학 및 암 치료 연구에 활용될 수 있으리란 기대다.
T2T 컨소시움을 공동 창립한 카렌 미가 산타크루즈대 교수는 "지난 20년 간 우리가 연구하지 못 했던 이 유전자 영역은 유전자의 작동 원리와 유전병, 인간의 다양성과 진화 등을 이해하는데 중요한 역할을 할 것"이라고 말했다.

■ 진화와 질병에 대한 새로운 접근 가능
실제로 새로 확인된 유전체 정보는 진화와 질병 연구에 대한 새로운 실마리를 보여준다. 연구진은 센트로미어 주변의 DNA 염기 서열을 파악해 인류 공통의 유인원 조상까지 거슬러 올라가는 연구를 할 수 있다는 가능성을 제시했다. 센트로미어의 중심부에서 멀어질수록 열화된 과거의 서열이 나타나고, 결국 먼 조상 유인원이 물려준 센트로미어가 나타나기 때문이다. 센트로미어가 유전자 안의 '화석층' 역할을 하는 것이다.
또 새 유전체 정보를 세계 각지의 사람 1600명의 센트로미어 DNA를 비교한 결과, 주요한 변이는 가까운 조상 중에 아프리카 사람이 있는 사람에게 주로 나타났다는 것도 드러났다.
이 정보를 인간이 아닌 유인원 염색체의 긴 서열들과 비교한 연구에서는 사람에게만 있는 부분들이 전두엽 발달과 시냅스 연결 강화 등 인간 고유의 특성을 보여주는 유전자와 관련 있다는 것이 밝혀졌다.
관련기사
- '차세대 유전체 합성' 등 10대 바이오 미래 기술 선정2022.02.28
- KAIST, 세계 최대 3차원 암 게놈 지도 구축2020.12.28
- 걱정되는 유전자 편집 부작용, 확 줄일 방법 찾았다2022.03.03
- 국제유가 100달러 육박…주간 상승률 43년만에 최고2026.03.07
연구진은 향후 한 개인의 유전체 분석을 넘어 여러 사람의 유전제 정보를 모아 분석하는 단계로 나아갈 계획이다. 이를 위해 T2T 컨소시움은 '인간 판지놈 리퍼런스 컨소시움'과 협업 중이다.