KAIST(총장 이광형)는 한동수 전기및전자공학부 교수 연구팀이 머신러닝을 활용, 기존 방식에 비해 속도를 3배 이상 높인 유전체 정렬 소프트웨어를 개발했다고 12일 밝혔다.
유전체 정렬 소프트웨어는 차세대 염기서열 분석 방법으로 생성한 유전체 조각 데이터를 온전한 유전체 정보로 조립하는데 쓰이는 소프트웨어다. 유전체를 무수히 많은 조각으로 잘라낸 후 각 조각을 참조 유전체에 기반해 조립, 유전체 정보를 해독한다. 조립된 유전체 정보는 암 등 각종 질병 예측과 맞춤형 치료, 백신 개발 등에 활용된다.
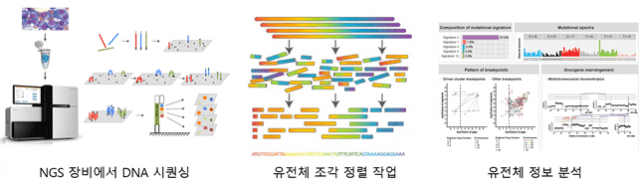
방대한 데이터를 빠르게 정확하게 처리해야 하기 때문에 강력한 연산 성능과 많은 비용이 소모된다. 지금까지는 미국 하버드대학과 인텔 등이 개발한 유전체 분석 도구 키트(GATK)를 기반으로 한 BWA-MEM이나 BWA-MEM2 등의 유전체 정렬 소프트웨어가 쓰였다.
한동수 교수 연구팀은 머신러닝 기반 인덱싱(색인) 기법을 유전체 정렬 소프트웨어에 처음으로 적용했다. 데이터의 분포를 머신러닝 모델이 학습, 데이터 분포에 최적화된 인덱싱을 찾는 방식이다. 유전자 조각 길이와 상관 없이 적은 연산량으로도 유전자 조각의 위치를 찾을 수 있다.
관련기사
- 인간 유전체 정보, 남은 퍼즐 조각 모두 찾았다2022.04.01
- '차세대 유전체 합성' 등 10대 바이오 미래 기술 선정2022.02.28
- '갤S25 엣지' 배터리·망원 지적에 삼성 임원 답은?2025.06.07
- '웨어러블 AI' 시장 뜨는데…글로벌 빅테크 "특화 메모리 없다" 지적2025.06.07
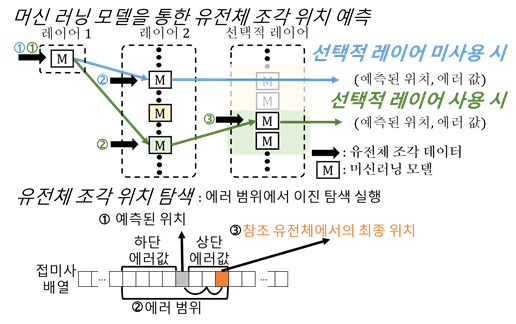
연구팀이 제안한 인덱싱 기법은 기존 인덱싱 기법보다 3.4배 정도 빨라졌고, 이로 인해 유전체 정렬 소프트웨어는 1.4배 가속화됐다. 한동수 교수는 "이번 연구를 통해 기계학습 기술을 접목해 전장 유전체 빅데이터 분석을 기존 방식보다 빠르고 적은 비용으로 할 수 있다는 것을 보여줬다"라며 "앞으로 인공지능 기술을 활용해 전장 유전체 빅데이터 분석을 효율화, 고도화할 수 있을 것"이라고 기대했다.
연구팀은 개발한 소프트웨어를 오픈소스로 공개했다. 관련 연구 성과는 학술지 '옥스포드 바이오인포메틱스(Oxford Bioinformatics)'에 게재됐다.