국내 연구팀이 코로나19의 RNA 전사체를 세계 최초로 분석하는 데 성공했다. 바이러스 전사체가 어떻게 구성됐는지 정확하게 확인할 수 있게 됨에 따라, 치료제 개발에도 속도가 붙을 것으로 전망된다.
기초과학연구원(IBS)은 김빛내리 서울대 생명과학부 교수가 이끄는 공동 연구팀이 코로나19의 원인인 사스코로나바이러스-2의 고해상도 유전자 지도를 완성했다고 9일 밝혔다.
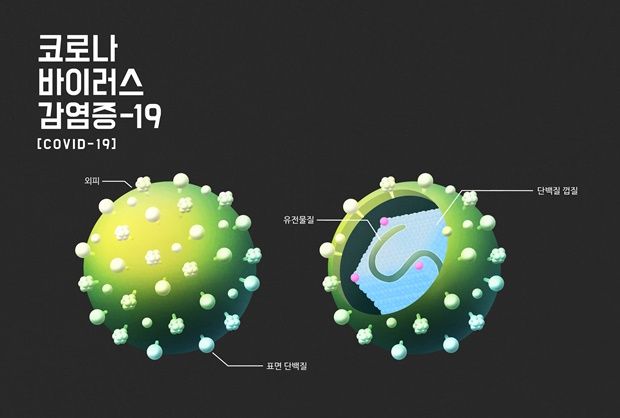
연구팀은 두 종류의 차세대 염기서열 분석법을 활용해 사스코로나바이러스-2가 숙주세포 내에서 생산되는 RNA전사체를 분석했다. 이 분석을 통해 바이러스 유전자의 정확한 위치를 찾아냈고 기존 분석법으로는 확인되지 않았던 RNA들을 찾았다. 그 결과 바이러스의 RNA 최소 41곳에서 화학적 변형이 일어난다는 사실을 확인했다.
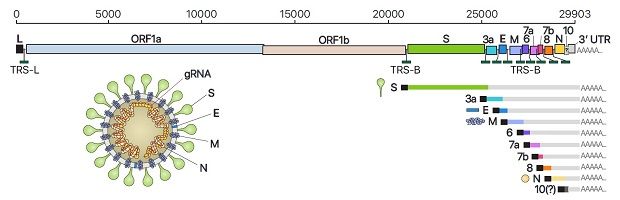
사스코로나바이러스-2는 DNA가 아니라 RNA 형태의 유전자를 지니고 있다. 바이러스는 숙주세포에 침투해 유전정보가 담긴 RNA를 복제하고, 유전체 RNA를 바탕으로 다양한 ‘하위유전체 RNA’를 생산한다. 하위유전체는 여러 단백질을 합성하고, 복제된 유전자와 함께 숙주세포 안에서 바이러스 완성체를 이룬 후 세포를 탈출해 새로운 세포를 감염시킨다.
기존 연구에서 사스코로나바이러스-2의 유전체 정보가 보고됐었지만, 이는 유전체 RNA 정보를 기반으로 유전자의 위치를 예측하는 수준에 불과했다. 김 단장 연구팀은 하위유전체 RNA를 실험적으로 규명하고 각 전사체의 염기서열을 모두 분석했다.
관련기사
- '코로나19' 저유가 시대…석유公, 비축유 늘린다2020.04.09
- 멀리서 체온 재는 로봇 등장 “코로나19 감염 우려 ↓”2020.04.09
- '함께 이겨내자' ADI, WHO 코로나19 기금 지원2020.04.09
- 코로나19에도 흔들림 없는 반도체 코리아2020.04.09
연구팀은 바이러스 RNA에서 메틸화와 같은 화학적 변형도 발견했다. IBS는 변형된 RNA들은 기존에 알려지지 않은 새로운 특성을 가질 수 있으므로, 이번에 발견한 변형은 사스코로나바이러스-2의 생활사와 병원성을 이해하는 데 도움이 될 것이라고 설명했다.
김빛내리 단장은 “이번 연구를 통해 사스코로나바이러스-2 각 전사체의 정량을 정확하게 파악했으며, 이를 토대로 진단용 유전자증폭기술(PCR)을 개선할 수 있을 것”이라며 “사스코로나바이러스-2 유전자에 대한 풍부한 정보와 세밀한 지도를 제시함으로써 바이러스의 증식원리를 이해하고 새로운 치료전략을 개발하는 데 기여할 것”이라고 말했다.