전자부품이 아니라 유기화합물인 디옥시리보핵산(DNA)을 데이터 저장매체로 사용할 수 있을까. 수년전만해도 연구자들 사이에서 실용성에 의문이 제기된 이 아이디어가 어느샌가 실제 구현 단계에 와 있다는 소식이 나왔다.

미국 지디넷은 지난 22일자 '실리콘은 잊어라 - SQL온DNA(SQL on DNA)가 데이터베이스(DB)를 위한 차세대 개척지'라는 기사를 통해, 전자식 컴퓨터의 DB 소프트웨어로 생화학모듈의 DNA에 데이터를 쓰고 읽는 최신 기술 동향을 소개했다.
기사는 현존 전자장치용 소재기술만으로는 충분한 데이터 저장과 계산 성능을 낼 수 없기 때문에, 사람들이 대체 가능한 데이터 저장매체를 찾아 왔다고 지적한다. 대다수 생명체의 유전정보를 저장하는 고분자 유기화합물 DNA를 그런 데이터 저장매체로 쓰는 것은 언뜻 이상하게 들릴 수 있지만, 실은 여러 이점을 얻을 수 있는 대안이라고 설명했다. 이 연구에서 최근 인기 오픈소스 DB인 포스트그레SQL(PostgreSQL)에 이런 DNA 스토리지를 통합할 수 있을만한 진전이 있었다고 덧붙였다.
기사 원문을 아래에 번역해 소개한다. [원문보기 ☞ Forget silicon - SQL on DNA is the next frontier for databases]
■ 정보 부호화 방법으로써의 DNA
시스템의 핵심은 DNA를 데이터 스토리지 레이어로 삼는 것이다.
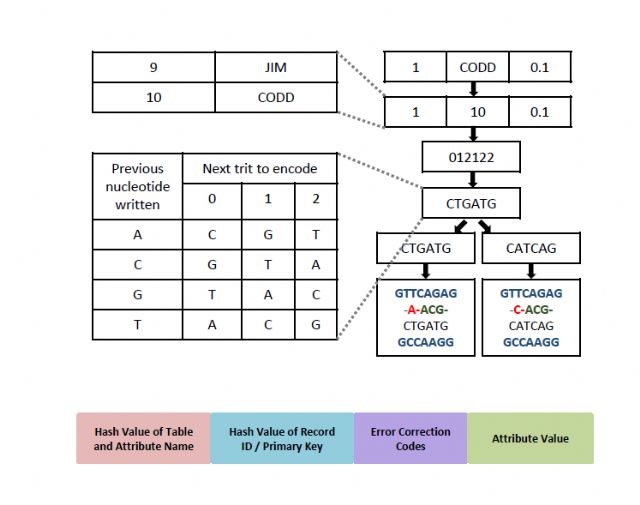
DNA는 '뉴클레오티드(nucleotide)'라는 단위물질이 여럿 연결돼 만들어진다. 뉴클레오티드는 염기, 당, 인산이 결합해 만들어진 분자다. 뉴클레오티드에 결합되는 염기는 아데닌(Adenine), 구아닌(Guanine), 시토신(Cytosine), 티민(Thymine), 네 종류다. 결합된 염기의 종류에 따라 뉴클레오티드도 네 종류로 나뉜다. DNA에서 네 종류의 뉴클레오티드 세 개가 '코돈(codon)'이라 불리는 그룹을 형성한다. 코돈은 우리 세포에 단백질 형성에 대한 지시를 내리는 단위다.
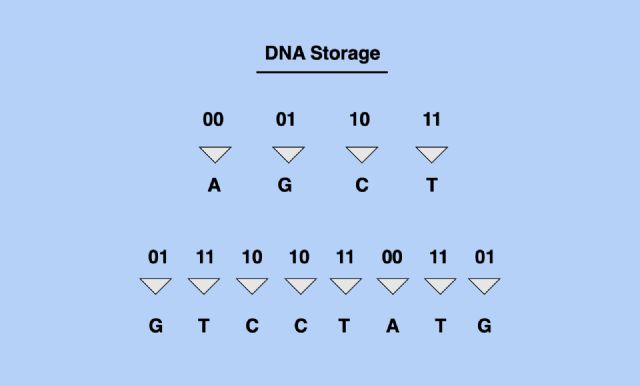
우리 IT인프라는 '0'과 '1'이라는 두 숫자를 담을 수 있는 비트(bits)에 정보를 저장한다. 반면 DNA의 정보는 앞서 언급된 아데닌, 구아닌, 시토신, 티민, 네 종류의 염기를 의미하는 기본 단위 문자열(A, G, C, T)로 저장된다. DNA에 비 유전정보를 저장하려면, 우선 이진값 데이터를 DNA의 구조에 맞는 AGCT 네 종류 문자 데이터로 변환해야 한다.
이론적으로는 전자기기가 0과 1 상태의 배열을 반도체나 디스크에 정보를 저장하듯 A, G, C, T의 배열을 저장하는 수단으로 DNA를 쓸 수 있다. 실제로는 어떨까.
분자 기술의 발전으로 이 방식이 부분적으로 실현 가능해졌지만 완벽하진 않다. 이론적으로 DNA에 저장하고 복원할 수 있는 방식으로, 정보를 인코딩해 실제 DNA 합성과 배열 조작을 수행하면 그게 작동한다는 얘기다.
앞서 마이크로소프트(MS)가 자동화된 DNA 데이터 저장 및 복원 시스템을 선보였다. 이는 시스템의 일부로 생산되는 합성 DNA 배열을 다룬다.
자연계의 DNA는 두 가닥의 뉴클레오티드가 만들어내는 이중나선형태로 구성된다. 이와 달리 데이터 저장용 DNA는 '올리고뉴클레오티드'라 불리는, 단일 뉴클레오티드 배열이다. 이는 DNA를 한 번에 하나의 뉴클레오티드로 조립하는 화학공정을 사용해 합성된다.
■ 현실 세계 데이터 저장에 DNA 사용하기
프랑스 유레컴(Eurecom) 데이터과학부의 라자 아푸스와미 조교수와 영국 임페리얼칼리지런던(ICL)의 스케일랩(SCALE Lab) 책임자 토마스 헤이니스가 '혁신데이터시스템리서치' 컨퍼런스에서 발표한 DNA 스토리지 관련 연구 논문이 눈길을 끈다. 논문은 '올리고아카이브, DBMS 스토리지 계층구조에 DNA 사용'이라는 제목으로 나왔다.
미국 지디넷 보도에 따르면 이들의 논문에서 수행한 연구는 DNA를 사용해 데이터를 저장하고 복원했다는 점에서 기존 아이디어를 계승했지만, 이 방법을 이미 상용화된 DB와 결합해 정형데이터를 저장하는 데 적용하고, 스토리지를 넘어 컴퓨팅을 구현했다는 점에서는 최초라고 한다.
DNA를 데이터 스토리지 레이어로 삼을 경우 실제로 기록 작업이 수행되면 매번 올리고뉴클레오티드가 합성돼야 한다. 이는 어떻게 작동할까.
실험실 기술자가 DNA를 매번 합성되게 만들고 그 화학공정 원료를 다시 채워넣어야만 하는 건 아니다. 아푸스와미와 헤이니스에 따르면, MS는 이미 자동화된 DNA 스토리지 기술을 구현하면서 DNA에 정보를 기록하는 합성 과정에 사람이 개입할 필요는 없음을 보여 줬다. 데이터센터의 IT인프라 운영에서 모든 세세한 작업이 사람의 감독아래 돌아가지 않듯이, DNA 스토리지 기반 데이터센터 역시 그럴 수 있다는 설명이다.

하지만 합성 DNA 배열을 사용한 스토리지가 당장 하드디스크 저장장치를 대체할 수준에 와 있는 것과는 여전히 거리가 멀다. 새로운 방식으로 데이터를 저장하려면 매우 느린 속도를 감수해야 한다. 처음엔 1메가바이트 데이터를 저장하는 작업에 1주일이 걸릴 정도였다. 여전히 아푸스와미와 헤이니스는 더 많은 연구가 필요하다는 점을 인정했다. 이제 기록 속도는 초당 수킬로바이트 정도로 상당히 개선됐지만 여전히 SSD보다 훨씬 느리다. 다만 연구자들은 이 정도의 속도라면 실제 아카이브 스토리지 목적으로 쓸 수 있을만한 수준이라고 봤다.
DB 엔진은 다양한 가격과 성능 특성에 따라 광범위한 장치의 3계층 조합을 사용한다. 성능 중시 계층의 데이터 저장 영역은 고성능 온라인분석처리(OLTP) 및 실시간 분석 애플리케이션을 통해 접근하는 데이터를 저장한다. 용량 중시 계층의 데이터 저장 영역은 지연시간을 크게 따지지 않는 배치 분석 애플리케이션을 통해 접근하는 데이터를 저장한다. 그리고 아카이브 목적의 계층은 보안 규제준수 확인과 법률적 감사 등 드물게 접근하는 데이터를 저장하는 데 사용된다. 일반적으로 이 계층에 사용되는 저장매체는 테이프라이브러리형 스토리지였다.
연구자들이 고안한 방식은 DB 스토리지 계층 구조에서 이런 테이프 스토리지를 DNA 기반 계층으로 대체할 수 있다. 합성 DNA는 특별히 세심한 절차를 사용해 보존되는데, 일반적인 장치 대비 DNA 기반 스토리지가 얼마나 효과적일는 아직 의문을 남긴다. 이 DNA 스토리지는 '올리고아카이브'라는 깃허브 프로젝트로 공개됐다.
■ SQL 온 DNA 실행
연구자들은 DNA 스토리지가 느리긴 하지만 병렬처리 용도에 큰 잠재력을 지녔다고 언급했다. 당장은 이 기술을 사용했을 때 고품질 스테레오 1분을 저장하는 데 10만달러 비용이 들지만, 장기적으로는 이보다 훨씬 더 풍부하고 저렴해 질거란 기대에서다. 규모에 따라서는 합성 DNA를 사용하는 스토리지 기술이 엄청나게 비싸지만 다른 스토리지 기술을 포함한 모든 과학적, 기술적 도약이 그래왔듯 이 기술의 비용도 크게 낮출 수 있을 것이라는 예상이다.
올리고뉴클레오티드같은 기술의 비용이 현실적인 수준으로 떨어진다면 병렬 동작하는 DNA 스토리지로 뭘 할 수 있을까. 일단은 전체적인 처리 속도를 높일 수 있다. 연구자들은 관계형 DB에 DNA 스토리지를 통합했고, 표준 DB 성능 벤치마크 지표중 하나인 TPC-H의 데이터와 쿼리를 가져와 포스트그레SQL 인스턴스에서 TPC-H 테스트를 수행했다. 모든 데이터에 직렬 액세스하는 방식이 아니라 선별 데이터 대상으로 진행한 테스트였다.
요컨대 이들을 통해 백엔드에서 DNA를 사용하는 DB 시스템에 정형데이터를 저장하고 이를 SQL로 질의하는 방식은 현실화됐다. 연구자들은 포스트그레SQL 시스템이 DNA에 관계형 데이터의 스키마 인식 인코딩 및 디코딩을 수행하는 아카이빙 및 리커버리 툴을 구축했다. 이 툴을 사용해 TPC-H의 테스트용 12킬로바이트짜리 DB를 DNA에 저장한 다음, 연산을 수행하고, 다시 복원했다. 이 DNA 스토리지는 데이터 일부에 선택적으로 접근해 그걸 처리하는 SQL 명령을 지원할 수 있다. DB에서 데이터를 가져오지 않고 명령을 수행할 수 있다. 아푸스와미와 헤이니스는 올리고뉴클레오티드 안에 SQL 조인(join) 구문과 같은 연산을 할 수 있는 방법을 발견했다. 생화학적 데이터 저장을 넘어 생화학적 데이터 연산을 수반한다는 얘기다.
연구자들은 DNA로 정보를 인코딩하고 디코딩하는 데 사용된 기술의 결함과 관련된 문제를 해결해야 했다. DNA상의 작업을 수행하려면 생화학적 조작에 알맞는 올리고뉴클레오티드를 생성하는 특수 인코딩 기법이 필요하다. DNA 데이터 읽기 작업은 현재 많은 오류를 유발한다. 기존 방식은 데이터 표현에 과도하게 의존했다. 데이터는 여러 복본으로 작성됐는데, 원본이 손상됐을 경우에 대비해 백업이 존재했던 것이다. 아푸스와미와 헤이니스는 이와 달리 메타데이터를 활용했다. DB 스키마 인지 기법을 활용해 블록에 데이터 비트를 추가하는 방식을 썼다. 이는 인코딩(쓰기) 작업 밀도와 디코딩(읽기) 작업 오류 식별성을 높이는 데 도움이 된다.
■ DNA는 데이터의 미래인가?
미국 지디넷은 "기술 스택 일부는 미숙하지만 이(연구)는 중요한 진전"이라면서 "이미 데이터센터용 중복 스토리지 확보가 게임체인저"라고 평했다.
아푸스와미와 헤이니스는 올리고아카이브 프로젝트에서 이 연구의 다른 부분을 맡아 준 UCA와 CNRS의 다른 연구자들과 협력하고 있다. 이들은 팀과 연구의 규모를 키울 수 있게 됐다. 유레컴, CNRS, ICL, UCA 그리고 DNA 합성기술 스타트업인 '헬릭스웍스'가 유럽연합(EU)으로부터 DNA 스토리지 후속 연구를 위한 예산을 확보했다. 이 시스템은 온전한 데이터 인코딩 자동화 사이클을 지원하고 그걸 DNA로 합성하며 배열로부터 다시 읽어들일 수 있도록 설계된다. 상이한 데이터 유형을 저장하고 스토리지 안에서 근거리 데이터 처리와 정확한 데이터 복원을 가능케 할 것이다.
관련기사
- "車가 서버이자 스토리지고 데이터센터인 시대"2019.07.31
- "AI 시대에 고성능 스토리지가 중요한 이유"2019.07.31
- 스토리지 업계, 데이터센터에 AI 심기 분주2019.07.31
- 尹 탄핵심판 선고 이틀 앞으로...과학기술계 입장은2025.04.02
연구자의 프로젝트는 EU의 미래신흥기술(FET) 정책기금으로 지원됐다. FET 정책기금은 급진적인 미래 신기술을 연구 주제로 삼으면서 해당 주제를 다루고자 하는 연구자가 적은 초기 단계 아이디어를 지원 대상으로 삼는다.
미국 지디넷에 따르면 아푸스와미와 헤이니스는 다른 연구자들, 특히 MS가 이 프로젝트에 비상한 관심을 보였다는 점이 주목할 만하다고 밝혔다. 이들은 프로젝트를 바라보는 다른 사람들의 태도에 대해 "2년전 사람들은 이 연구가 너무 먼 얘기라고 무시했지만 오늘날 우리가 작업하고 있는 것을 얘기하면 '더 들려 달라'고 말한다"고 언급했다.